インフルエンザウイルスのゲノム合成過程を観察することに成功!
金沢大学ナノ生命科学研究所(WPI-NanoLSI)の福田真悟特任助教,安藤敏夫特任教授(特別功績教授)とスペインのIMDEA ナノサイエンス研究所のボーハ・イバーラ准教授,ナショナルバイオテクノロジーセンター(CNB-CSIC)のハイメ・マーティン=ベニート博士らの共同研究グループは,高速原子間力顕微鏡(高速AFM(※1))を用いてA型インフルエンザウイルスのゲノムが合成される様子を世界で初めて撮影することに成功しました。
インフルエンザは,インフルエンザウイルスに感染することによって起こる呼吸器感染症です。インフルエンザウイルスは,単独で増殖することはできず,感染した細胞の中で遺伝子のコピーを作り増殖していき,感染したほとんどの細胞を死滅させます。一方で,一度インフルエンザウイルスに感染すると体内で抗体が作られ免疫を獲得できます。ワクチンによる予防接種もこの仕組みを利用したものです。しかしながら,インフルエンザウイルスの遺伝情報は,増殖時に変異の導入が起こりやすく,獲得した免疫では感染を防ぎきることができなくなり,人に対して強い感染力を持つように変異すると,新型インフルエンザウイルスとなりパンデミックを引き起こします。この変異の導入は,ウイルスRNAポリメラーゼによるゲノム(遺伝情報)の写し間違えが要因の一つと考えられています。そのため,どのようにしてウイルスRNAポリメラーゼが遺伝情報を読み取り,コピー(RNA)を合成するかを明らかにすることは,抗ウイルス薬などの薬剤開発につながると考えられます。
本研究では,高速AFMを用いてゲノム合成の基本装置であるvRNP(※2)を可視化することによって,ウイルスRNAポリメラーゼがゲノムを読み取りコピーを合成する様子を観察することに成功し,その分子メカニズムを明らかにしました。本研究の成果は将来,インフルエンザウイルスの治療薬やワクチンの開発の指針として活用されることが期待されます。
本研究成果は,2024年7月16日(米国東部時間)にアメリカ化学会が発行する英文誌『ACS Nano』のオンライン版に掲載されました。
研究の背景
A型インフルエンザウイルスは,8本の分節した1本鎖RNAをゲノム(遺伝情報)として持ち,それぞれのRNAはウイルス由来のRNA依存性RNAポリメラーゼと複数のNPタンパク質からvRNPを構成します(図1 (A))。RNAはNPに巻き付き,RNAポリメラーゼは合成の開始地点であるプロモーター領域を含むRNAの両末端に結合しています。これまでの研究から,RNAポリメラーゼはプロモーター領域から解離することなくRNAを合成することが明らかになっています。しかしながら,どのようにしてRNAポリメラーゼが,vRNP複合体の中でNPに巻き付いているRNAの遺伝情報を読み取り,コピーを合成するのかは明らかになっていませんでした。また,分子レベルでvRNPによる転写や複製反応を可視化した研究が存在しないため,ウイルスRNAの合成にどれくらいの時間がかかるのか,またRNAポリメラーゼは連続して複数回のRNAの合成を行うことができるのか,といったインフルエンザウイルスの増殖力に関わる重要な事柄も明らかになっていませんでした。
研究成果の概要
本研究では,遺伝子操作技術を用いてウイルスRNAを短く切断した再構成vRNPを精製し,高速AFM観察を行いました。ネイティブなvRNPでは,RNAポリメラーゼはRNAの合成中にvRNPの内部に存在しているため,その動きを可視化することが困難でした (図1 (A))。一方で,再構成vRNPは特徴的なリング構造を持ち,RNAポリメラーゼを容易に特定することが可能です(図1 (B))。
高速AFM観察中にNTP(※3)を加えると,vRNPは背の高さが約2倍になりリング形状から大きく構造を変化させ,数秒後に元のリング形状に戻る様子が観察されました(図1 (C))。さまざまなNTP濃度で撮影を行うと,NTPの濃度に応じて構造変化の時間が変わることが分かりました。高NTP濃度(~1 mM)では,RNAの合成速度は約30ヌクレオチド/秒で,この結果は最も長いウイルスRNA(2200 ヌクレオチド)を約1分間で合成することが可能なことを示しています。さらに,元のリング構造に戻ったvRNPが,同様の構造変化を繰り返し行う様子も観察されました。
この結果から,vRNP内でRNAポリメラーゼが連続的にRNAの合成を行うことができる直接的な証拠を示すことができました。さらに,合成した新生RNAの高次構造を不安定化させるNTPアナログを用いて観察を行うと,RNAの合成速度が速くなることが分かりました。反対に,新生RNAの高次構造を安定化させるNTPアナログを用いるとRNAの合成速度は低下しました。この結果は,新生RNAによってRNAの合成が調整されることを示唆しています。
本研究で明らかになったvRNPの構造ダイナミクスは,ネイティブなvRNPの電子顕微鏡観察から提案されていた転写モデルとよく一致しており,ネイティブなvRNPにおいても同様のメカニズムでRNAの合成が進行することが考えられます。
今後の展開
本研究では,ウイルス由来のRNAポリメラーゼがvRNP内でRNAを合成する分子メカニズムを明らかにしました。本研究で用いた再構成vRNPは観察基板と干渉することなく,その構造動態を可視化することができるため,高速AFM観察に適しています。今回の高速AFM観察で新生RNAの高次構造が合成速度を調整することが明らかになりましたが,合成されているRNAを可視化することはできませんでした。そのため,どのように新生RNAとRNAポリメラーゼが相互作用し,RNAの合成を調整しているのかは明らかになっていません。
今後,高速AFMの高時間・空間分解能化や蛍光顕微鏡との複合計測を行うことによってvRNPの構造変化とRNAの伸長過程を同時に可視化することも可能になると考えられます。また,本研究で確立した実験システムを応用することで,cRNAを鋳型として子孫ウイルスRNAが合成される様子も可視化できると考えられます。これらの計測によって,インフルエンザウイルスゲノムの複製・転写過程のさらなる詳細なメカニズムが明らかになることが期待されます。
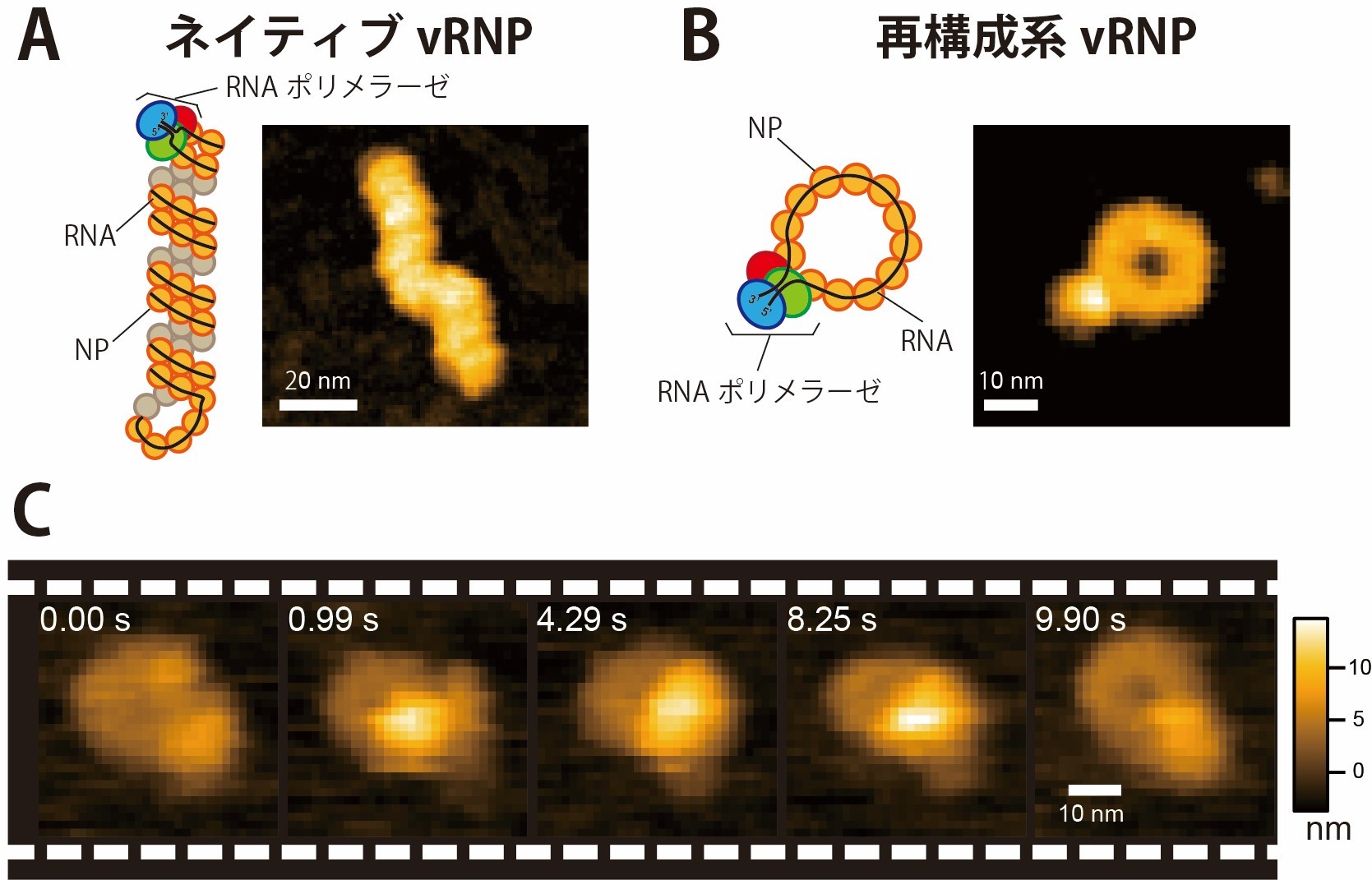
図1 (A) ネイティブなvRNPの高速AFM画像。(B) 再構成系のvRNPの高速AFM画像。(C) NTPを加えた後の再構成系vRNPの高速AFM連続画像。0.99秒の時に,大きく構造変化してvRNPの背が高くなり,9.90秒の時にRNAの合成を終えて元のリング構造に戻る様子が観察された。
用語解説
掲載論文情報
- 論文タイトル
- Conformational dynamics of influenza A virus ribonucleoprotein complexes during RNA synthesis (インフルエンザウイルスのゲノム合成マシナリーの構造ダイナミクス)
- 著者
- Diego Carlero, Shingo Fukuda*, Rebeca Bocanegra, Toshio Ando, Jaime Martin-Benito*, Borja Ibarra* (ディエゴ・カルレロ,福田真悟*,レベッカ・ボカネグラ,安藤敏夫,ハイメ・マーティン=ベニート*,ボーハ・イバーラ*) *Corresponding authors
- 掲載誌
- ACS Nano
- 掲載日
- 2024.07.16
- DOI
- 10.1021/acsnano.4c01362
- URL
- https://pubs.acs.org/doi/10.1021/acsnano.4c01362

