天然変性タンパク質の動的かつ多様な構造の定量的解析に成功 ~天然変性タンパク質の理解に新しい道を拓く~
金沢大学ナノ生命科学研究所の安藤敏夫特任教授,古寺哲幸教授らは,高速AFMイメージングにより,天然変性タンパク質(IDP)の動的構造を高い精度で解析することに成功しました。
研究の背景
一定のアミノ酸配列を持つポリペプチド鎖は特定の構造に折れ畳まれ(フォールドして)タンパク質となり,一つの機能を担うという常識は,約20年前に発見されたIDPの存在により覆されました。αヘリックスやβシートといった二次構造から,さらに三次元構造へとフォールドしたタンパク質では,その構造が機能を理解するための基礎であることから,X線回折,電子顕微鏡,核磁気共鳴(NMR)といった手法によって多くのタンパク質の構造が決定されてきました。IDPでも構造は機能と密接に関係しています。しかし,分子全体にわたり,あるいは,分子内の大きな領域で二次構造をとらないIDPでは常に構造が大きく揺らいでいるため,構造解析は非常に困難です。IDPは結晶化しないためX線回折を用いることができず,解けた(アンフォールドした)領域(IDR)は細すぎて電子顕微鏡では可視化できません。NMRはIDP内の秩序構造領域の構造解析とIDRの位置決定に有効ですが,完全にアンフォールドしているのか緩くフォールドしているかなどの情報を得ることができません。NMRが与える局所構造情報を補い,分子全体の広がりや形の情報を得るためにX線小角散乱(SAXS)が利用されています。しかし,NMRもSAXSもアンサンブル平均手法であるため,互いの間を動的に遷移するそれぞれの構造を抽出することも,それらの割合を決めることもできません。このように,姿・形とその変化を明確に捉えられないという問題があるため,IDPの理解は従来型のタンパク質に比べ非常に遅れています。IDPがタンパク質世界の約半分を占め,さまざまな生命現象に重要な役割を担っていることを考えると,IDPの構造解析に有効な手法を見いだすことは生命科学全体にとって非常に重要な課題です。

(左)安定した秩序構造をとる従来型のタンパク質、(右)安定した秩序構造をとらない天然変性タンパク質
研究成果の概要
金沢大学のグループが開発した高速AFMは,タンパク質分子の構造と動作を可視化できることから,秩序構造をとるタンパク質の機能中の動的プロセスの観察に広く用いられ,従来技術では不可能なメカニズムの発見に貢献してきました。本研究では,高速AFMが,構造が大きく揺らぐIDPの構造解析にどこまで有効であるかを探りました。分子全体がアンフォールドしたIDPよりも,秩序構造部分とIDR(ひも)から成るIDPの方が多いことが知られています。NMRは秩序構造部分の詳細構造とひもの位置を特定できるものの,それが与える描像はIDPが「球」と「ひも」でできているといった単純なものでした。この描像を高速AFM解析により,さらに具体的に写実的に記述できるかが重要なポイントでした。
まず,NMRなどの手法で球とひもの位置がアミノ酸レベルでほぼ決定されているIDP(PQBP-1)を高速AFMで観察しました。PQBP-1はひも1本とその端にある球1つからなり,球の大きさは変わらず,ひもは常に太さが約0.5 nmのひもとして観察されました。これを反映して,大きく揺らぐひもの両端距離R2Dの分布は単一のピーク(平均両端距離 <R2D>)を示しました。そこで,ひもを短くした4種のPQBP-1変異体の<R2D>値を測定し,合計5個の<R2D>値とそれぞれのひもに含まれるアミノ酸数Naaとの関係を調べたところ,<R2D> = 1.18 (nm)×Naa0.51というべき乗則が成り立っているようでした。5種類のPQBP-1のひもに含まれるアミノ酸の成分は異なっていても,<R2D>とNaaの間に一定の関係があるという意外な事実を示唆しています。さらに,このべき乗則の普遍性を調べるべく,球とひもの位置がアミノ酸レベルでほぼ決定されている他のIDP(Atg1, Atg13, NTAIL, FACT)についても高速AFMを用いて観察しました。その結果,NMRが与える描像よりもかなり複雑で,球とひもとの結合解離や,球が解けて小さい球になり再び大きな球に戻るといった振舞いが観察されました。これらの振舞いを反映して,R2Dの分布には二つのピークが現れました。短い方の<R2D>値はイオン条件に依存して変化しますが,長い方の<R2D>値は一定でした。長い方の<R2D>を採用し,PQBP-1のデータと合わせてみると,上記のべき乗則より精度の高い<R2D> = 1.16 (nm)× Naa0.52という関係が得られました。このべき乗則は解けたひもに対する普遍則と考えられます。このことは非常に重要で,アミノ酸成分と配列がまったく異なる場合でも,太さ0.5 nmのひもとして観察されれば,その両端距離の平均値からそこに含まれるアミノ酸数を決定できます。アンサンブル平均手法では,異なる状態を分けて観察することができませんが,高速AFMでは個々の状態を区別して観察できるため,このべき乗則を有効に利用できます。
そこで,このべき乗則を利用して,NMRではいまだ十分に特定できないIDP(PNTとSic1)の構造を高速AFMで解析しました。どちらのIDPもひもはひものままでいることはなく,フォールドした状態とアンフォールドした状態を行き来していました。アンフォールドした状態に上記のべき乗則を適用し,両状態の構造を解析した結果を図1b,cに示します。図1aはNTAILについてのもので,NMRの結果と一致します。PNTの結果もNMRの結果に一致しますが,N端側のαヘリックスとひもがまとまった領域が存在するというNMRでは検出されていない事実に加え,この領域と負電荷に富んだひもが結合解離するという新事実が明らかになりました。このまとまった構造の存在は,N端から99残基までの領域はタンパク質分解酵素で分解されないという生化学的事実と一致し,高速AFMの構造解析の精度の高さを物語っています。緩くフォールドするSic1についてはこれまで有効な手法がなかったため比較すべきデータは皆無ですが,この精度の高さから判断して,図1cに示す結果は信頼できると判断できます。
他の手法では調べることができないIDPの状態間の構造遷移の速さを高速AFMのデータは与えることができます。ひもの長さ変化や球の高さ変化の時系列データを解析すると,図1に示すように状態間の遷移速度を決定することができます。構造についても遷移速度についてもここまで詳細かつ定量的にIDPの姿を記述する方法は他にはありません。
今後の展開
NMRで得られる局所構造情報と高速AFMで得られる種々の情報を組み合わせると,片方だけからの情報と比べはるかに詳細なIDPの構造情報が得られることがはっきりしました。この融合手法を利用すれば,IDPの構造解析は各段に進展し,それによりIDPの理解が加速されるものと期待されます。現在金沢大学と米国の大学との間で,本研究で開発した手法を活用して,病気の原因となるIDPの複数の変異体の構造解析を進めています。IDP内の遠く離れた部位間での相互作用が変異に影響されることや,秩序・無秩序状態間を遷移する領域の構造安定性と生理活性との間に相関があることなどが分かりつつあります。病気の原因となるIDPの構造異常を可視化できるようになったことから,病理の分子レベルでの理解が深まるだけでなく,構造異常を元に戻すような化合物の開発も可能になることが期待されます。
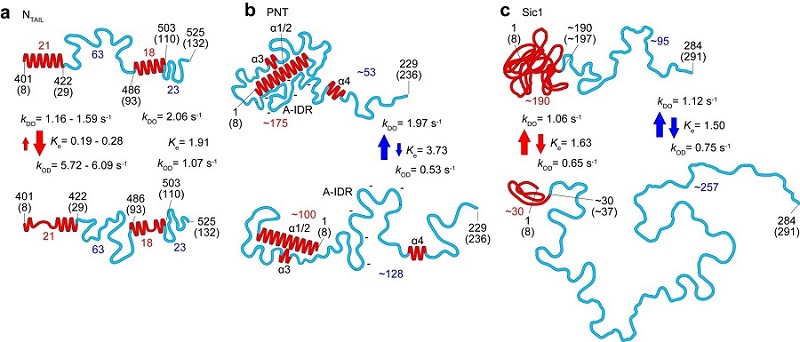
図1.高速AFM解析で得られた三種のIDPの構造とダイナミクス。図中の赤で示した部分はαヘリックスないしは緩くフォールドした構造を,水色で示した部分はフォールドした構造をとっていない天然変性領域を示す。上のパネルはフォールドした構造状態を,下のパネルはアンフォールドした構造状態を示す。赤字の数値はフォールドした構造に含まれるアミノ酸数を,青字の数値はアンフォールドした構造に含まれるアミノ酸数を示す。赤の矢印は高さの変化から判断される遷移を,青の矢印はアンフォールドした構造の両端距離の変化から判断される遷移を示す。Keの値はフォールドしやすさを示す。kODはフォールド状態からアンフォールド状態への遷移速度を,kDOはアンフォールド状態からフォールド状態への遷移速度を表わす。
用語解説
掲載論文情報
- 論文タイトル
- Structural and dynamics analysis of intrinsically disordered proteins by high-speed atomic force microscopy (天然変性タンパク質の構造とダイナミクスの高速AFMによる解析)
- 著者
- Noriyuki Kodera1, Daisuke Noshiro1, Sujit K. Dora2, Tetsuya Mori3, Johnny Habchi4, David Blocquel4, Antoine Gruet4, Marion Dosnon4, Edoardo Salladini4, Christophe Bignon4, Yuko Fujioka5, Takashi Oda6, Nobuo N. Noda5, Mamoru Sato6, Marina Lotti7, Mineyuki Mizuguchi8, Sonia Longhi4,* and Toshio Ando1,*
(古寺 哲幸,能代 大輔,Sujit K. Dora,森 哲哉,Johnny Habchi,David Blocquel,Antoine Gruet,Marion Dosnon,Edoardo Salladini,Christophe Bignon,藤岡 優子,小田 隆,野田 展生,佐藤 衛,Marina Lotti,水口 峰之,Sonia Longhi,安藤 敏夫)- 金沢大学ナノ生命科学研究所
- 金沢大学理工研究域数物科学系
- 金沢大学大学院自然科学研究科数物科学専攻博士前期課程
- エクス=マルセイユ大学(Aix-Marseille University)・フランス国立科学研究センター(CNRS, Laboratoire Architecture et Fonction des Macromolecules Biologiques (AFMB))
- 微生物化学研究所
- 横浜市立大学大学院生命医科学研究科
- ミラノ=ビコッカ大学バイオテクノロジー・バイオサイエンス学科(Department of Biotechnology and Biosciences, State University of Milano-Bicocca)
- 富山大学薬学部
- 掲載誌
- Nature Nanotechnology
- 掲載日
- 2020.11.23
- DOI
- 10.1038/s41565-020-00798-9
- URL
- https://www.nature.com/articles/s41565-020-00798-9
Funder
詳しくはこちら

