高速AFM像から原子レベルで生体分子の動きを再現する新しいモデリング技術の開発に成功
金沢大学ナノ生命科学研究所(WPI-NanoLSI)のホルガー・フレクシグ特任准教授、名古屋大学トランスフォーマティブ生命分子研究所(WPI-ITbM)/名古屋大学大学院理学研究科/理化学研究所計算科学研究センター(R-CCS)のフロハンス・タマ教授、理化学研究所計算科学研究センター(R-CCS)の宮下治上級研究員を中心とする共同研究チームは、高速原子間力顕微鏡(高速AFM)(※1)で得られた実験データをもとに、生体分子がどのように動くのかを原子レベルで理解するための、新しい解析手法(融合的モデリングワークフロー)の開発に成功しました。
高速AFMは、生体分子の動作を直接観察できる唯一の技術ですが、空間分解能の制約により原子レベルでの理解には限界がありました。本研究では、AFMトポグラフィ画像からタンパク質の動的な三次元原子構造モデルを推定できる新しい計算フレームワークが開発されました。この手法は、既知の静的構造を基に構造変化を推定し、実測画像に最も一致する原子モデルを再構築するものです。さらに、AFM画像解析ソフトウェアに実装することで、実測データから直接、原子レベルの構造ダイナミクスを推定できるようになりました。さらに、本手法は非常に巨大なタンパク質集合体にも適用可能であり、それらのタンパク質集合体についても高速AFM動画から原子レベルの動的分子動画を再構築することに成功しました。
ここでの開発は将来、私たちの体の中で日夜行われている、生物学的プロセスをナノスケールで解明することに広く活用されることが期待されます。
本研究成果は、2025年9月18日(現地時間)に米国科学誌『ASC Nano』のオンライン版に掲載されました。
【研究の背景】
高速原子間力顕微鏡(高速AFM)は、タンパク質などの生体分子が動作する様子を直接観察できる唯一の計測技術です。しかし、高速AFMは空間分解能に制限のある表面走査型の技術のため、分子機能を原子レベルで理解するには十分な情報が得られないという課題があります。これまでにもこうした限界を克服するための計算モデリングのアプローチが試みられてきましたが、実測データから原子レベルの情報を再構築することに成功した例はほとんどありませんでした。
【研究成果の概要】
今回、ホルガー・フレクシグ特任准教授(金沢大学WPI-NanoLSI)とフロハンス・タマ教授(名古屋大学WPI-ITbM/大学院理学研究科/理化学研究所)を中心とする研究チームは、AFMトポグラフィ画像からタンパク質の動的な三次元原子構造モデルを推定できる新しい計算フレームワークとそのソフトウェア実装を発表しました。この手法ではタマ研究グループが新しく開発した、計算効率の高い「フレキシブル・フィッティング(※2)」の手法を採用しています。これは既知の静的構造情報を基に、起こりうる構造変化を推定することで、実測AFM画像に最もよく一致する原子レベルの分子構造モデルを再構築するものです。そして、この手法をフレクシグ研究グループが開発・運用しているソフトウェアBioAFMviewer(※3)に実装し、実測されたAFM画像データを直接解析できるワークフローを実現しました。
本手法を用いて、WPI-NanoLSIの古寺哲幸教授および紺野宏記准教授の実験グループが取得したいくつかのタンパク質の高速AFM画像データを解析した結果、フレキシブル・フィッティングにより、大きな振幅運動を含む原子レベルモデルの推定が可能であることが明らかになりました。これにより、分解能に制約のある測定データからでも、機能的構造ダイナミクスをより正確に把握できるようになります。さらに、約 28万個の原子で構成される4メガダルトンのアクチンフィラメントを対象とした解析では、BioAFMviewer による高効率フレキシブル・フィッティングが非常に大型なタンパク質集合体にも適応可能なことを実証しています。さらに顕著な成果としては、高速AFMトポグラフィの動画データから、機能に関わるタンパク質構造ダイナミクスを原子レベルの分子動画として再構築することにも成功しました。
【今後の展開】
本研究により、高速AFM実験データと分子モデリングとを融合する新しい解析手法の開発が進められました。計算効率の高いフレキシブル・フィッティングを独自のソフトウェアに実装することで、単一分子イメージングデータの大規模解析が可能になり、高速AFMの解析能力を最大限に活用することができるようになります。これにより、ナノスケールでの生物学的プロセス解明への応用が広がることが期待されます。
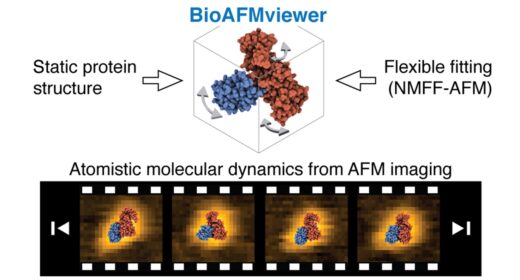
高速AFMイメージングデータから原子スケールで生体分子ダイナミクスを推定するためのBioAFMviewerフレキシブル・フィッティングの概略図(画像は元論文よりCC BY-NC-ND 4.0ライセンスで使用)
用語解説
BioAFMviewerには、生体分子の可視化のための統合分子ビューア、対応するAFM画像シミュレーター、さらに多彩な解析ツールボックスが組み込まれており、AFM実験データを直感的に解析できるユーザーフレンドリーな環境を実現しています。また、統合されたNMFF-AFMフレキシブル・フィッティング手法は、GPU(グラフィックカード)による並列計算によって性能が大幅に向上しています。BioAFMviewerソフトウェアは、プロジェクトウェブサイト www.bioafmviewer.com から無償でダウンロード可能です。
掲載論文情報
- 論文タイトル
- Flexible Fitting to Infer Atomistic-Precision Models of Large-Amplitude Conformational Dynamics in Biomolecules from High-Speed Atomic Force Microscopy Imaging (高速原子間力顕微鏡イメージングで得た生体分子の大きな構造動態に関する原子レベル精度モデルを推定するためのフレキシブルフィッティング法)
- 著者
- Romain Amyot, Osamu Miyashita, Xuan Wu, Kazusa Takeda, Noriyuki Kodera, Hiroki Konno, Florence Tama, Holger Flechsig (ロメン・アミヨ、宮下 治、ウー・シュアン、武田 春冴、古寺 哲幸、紺野 宏記、フロハンス・タマ、ホルガー・フレクシグ)
- 掲載誌
- ACS Nano
- 掲載日
- 2025.09.18
- DOI
- 10.1021/acsnano.5c10073
- URL
- https://pubs.acs.org/doi/10.1021/acsnano.5c10073

